하지만, 1990년대 후반 크레이그 벤터( J. Craig Venter )가 셀레라(Celera genomics)사를 설립 대규모 자동 게놈 해독기를 장착하고 shortgun sequencing이라는 초고속 게놈 해독법을 이용해 인간 게놈을 분석하면서 이 지루한 프로젝트는 흥미진진한 소설 같은 상황으로 전개가 된다.
셀레라의 분석 기술로는 정부 주도의 다국적 팀에 의해 10년 이상이 소요될 것으로 예측되는 프로젝트를 불과 2-3년 안에 완료 할 수 있었고, 미생물 게놈을 빠른 시간 안에 분석 해 발표하면서 이는 기정 사실화 되었다. 정부 주도 하의 다국적 팀에게 이는 엄청난 위협이었고, 위기감을 불러 일으켰다. ( 과학자의 입장에서 보자면 수년간 노력한 일이 수포로 돌아가는 것과 마찬가지인데, 과학 연구에서 2등은 큰 의미가 없기 때문. 이와 관련한 스토리는 벤터의 자서전 Life decoded에 자세히 기록되어 있는데, 정부 주도 팀의 관료적 행태들은 구역질이 날 정도의 장면들이 많다. )
Shortgun genome sequencing
Shortgun sequencing 은 인간의 긴 30억개 염기 서열을 평균 500-1,000 개 염기 길이의 작은 단위로 잘라 읽은 후, 각각을 퍼즐 맞추기 식으로 이어 붙여 전체 30억개 염기 서열 부위를 완성하는 형태다.
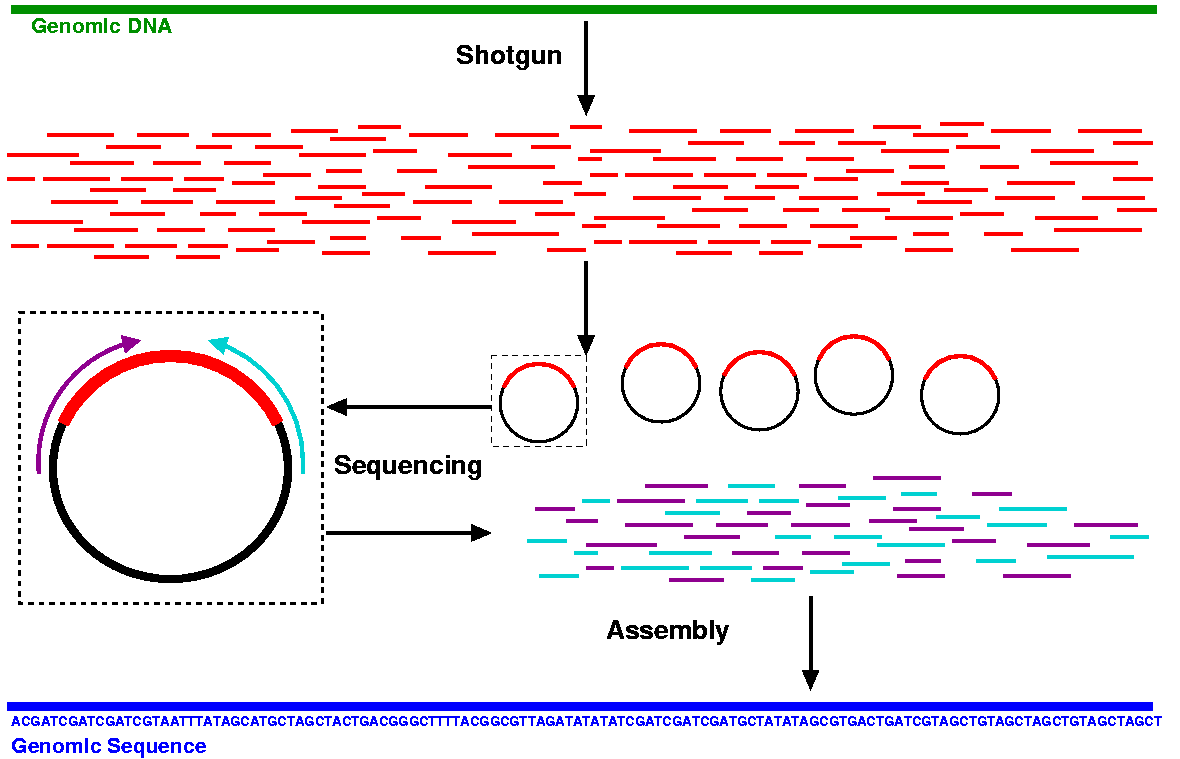 |
| Shortgun sequencing 방법 모식도 |
Shotgun sequencing 방법은 얼핏 매우 간단해 보이지만, 이 방법은 Bioinformatics 가 없이는 애초에 불가능한 서열 분석 방법이었다.
Shortgun sequencing을 통해 예를 들어 세개의 염기 서열 조각이 아래와 같이 주어진다고 하면 어떻게 이어 붙여야 할까?
AGTGT, GTAAC, ACTAG
아래와 같이 이어 붙이면 된다.
AGTGT
GTAAC
ACTAG
--------------------------
AGTGTAACTAG
아래와 같은 방식도 가능하다.
ACTAG
AGTGT
GTAAC
--------------------------
ACTAGTGTAAC
어떤 쪽이 옳은 것일까? 이런 서열 조각이 수천만개 있다면 어떨까? 어떻게 이 조각들을 제대로 꿰어 맞출 수 있을까? 이는 굉장히 정교한 수학적 모델을 필요로 했으며, 엄청난 컴퓨팅 파워를 필요로 하는 문제로 정통 생물학자들이 해결할 수 있는 문제가 아니었다.
이 문제를 푸는데 적용할 만한 수학적 이론은 Shortest common superstring problem (SSP), Eulerian graph problem 등이 있었고, Genome sequencing에 적용할 만한 이론들로 Michael Waterman 등의 기여로 대체로 정립,개발되어 왔지만, 실제로 이를 대량의 염기 서열 조각을 이어 붙이는 시스템에 적용하여 제작된 전례가 없었다.
생물정보학자 전쟁 :: Eugene Myers vs. Jim Kent
Eugene Myers 를 필두로 하는 Bioinformatics 팀이 셀레라 측의 Shortgun sequencing을 위한 염기 서열 조각 맞추기 문제를 위한 시스템을 성공적으로 완성했다. Gene Myers라고 흔히 불리는 Eugene Myers 는 NCBI에서 1990년 지금까지 가장 광범위하고 성공적으로 사용되는 생물학 프로그램 BLAST 를 공동 개발한 Bioinformatics 초기 영웅 중 한명이었는데, 그가 Celera에 합류하여 라떼 커피를 친구삼아 밤낮 가리지 않고 일에 몰두한 덕에 비소로 셀레라의 Shortgun sequencing 기술이 완성될 수 있었다.
Shortgun sequencing 시스템을 하드웨어적, 소프트웨어적으로 완벽히 갖추고 인간 게놈 분석의 막바지에 다다르고 있었던 셀레라 사에 비해, 다국적팀( 인간게놈컨소시움)은 정부의 중재로 셀레라 사와 함께 인간 게놈 분석 초안을 발표하기로 약속한 몇달 전이 되기 까지 이 게놈 조각 퍼즐 맞추기( Sequence assembly ) 를 위한 시스템이 완성되지 않아 초조하게 발을 구르는 상황이었다.
10년의 노력, 3조원의 정부 연구비를 정당화 하기 위해서 이 시스템은 너무나 간절했던 것이었다. 이 때, UCSC 생물학과에서 박사과정 학생이던 Jim Kent (William James Kent )가 David Haussler 와의 협력 하에 학교에서 새로 구매한 50대의 컴퓨터를 리눅스 클러스터로 사용하여 한달 만에 이 시스템을 완성한다. Haussler와 협력이라고 하지만, 이 프로그램의 코드는 100% Kent 혼자서 한달 동안 밤낮없이 달려들어 완성한 것이었다. Kent가 완성한 이 프로그램을 이용해 비로소 다국적팀도 셀레라사에 뒤지지 않게 성공적으로 전체 인간 게놈 지도를 완성할 수 있었다. ( 정확히는 Kent의 프로그램을 이용한 다국적 팀이 3일 빨리 완성)
Kent가 생물학과 박사과정 학생이었지만, 사실 그는 3D graphics 엔진을 개발하는 컴퓨터 프로그래머로 일하던 전문 프로그래머였는데, 생물학에 매력을 느껴 community college에서 생물학 과목을 수강한 후, UCSC 생물학과 박사과정에 늦깍이 진학을 한 특이한 이력을 가지고 있던 학생이었다.
이 때의 성과를 바탕으로 UCSC는 UCSC genome browser라는 웹 기반 게놈 검색/분석 시스템을 서비스하게 되는데, 현재 이는 BLAST와 마찬가지로 생물학자들에게 매우 일상적으로 이용되는 Bioinformatics 서비스로 자리 메김했고, 덩달아 UCSC 는 그저그런 UC 계열 대학에서 세계 최정상의 게놈 연구 기관 중 한 곳으로 자리 메김 하게 되었다.
( Kent의 사례는 왜 다양한 background를 가진 직원/학생들을 창의적 업무를 요하는 회사/대학에서 뽑아야 하는지를 극명하게 보여주는 또하나의 좋은 예가 된다 )
Bioinformatics on the go
Bioinformatics 라는 분야는 Computational biology 라고 명명된 좀 더 수학적 이론에 치우친 분야를 토대로 게놈프로젝트 이전 1970년대 부터 이미 존재해 온 분야였지만, 인간게놈프로젝트를 기점으로 그 필요성과 역할이 크게 확대되었으며, 이에 대한 수요도 크게 늘어나게 되었다.
인간게놈프로젝트를 기점으로 비로소 독립된 Bioinformatics 교육 프로그램들이 미국의 대학들에 설립되기 시작했고( 연구 조직이 있었던 곳은 꾀나 있었지만, 독립적 학위 과정 프로그램을 가진 곳은 없었다), Michael Waterman이 1982년 부터 그 기틀을 잡아왔던 USC, 인간게놈프로젝트를 통해 유명해진 David Haussler 가 중심이 된 UCSC, 그외 Boston University 가 초기 독립된 Bioinformatics 학위 과정을 대학에 개설했는데, 이중 UCSC는 학부생 레벨( Bachelor degree)의 코스를 유일하게 설립해 운영을 시작했다.
국내에서는 2001년 숭실대학교에 최초의 학부 과정 학과가 개설(현재는 의생명공학부)되었고, 부산대학에 최초의 대학원 과정이 개설되었다. 2002년에는 서울대에 생물정보협동과정 대학원 과정이 개설되었고, 2003년에는 정문술 회장의 300억 기부로 KAIST에 Biosystems 이라는 이름으로 Bioinformatics 를 교육하는 학과(현재는 Bio & Brain Engineering 학과)가 만들어졌다.
인간게놈프로젝트에서 극명하게 드러난 것은 생물학이 이제 Large scale data 생산하는 시대에 접어들었고, 생물학이 이 large scale data의 분석을 통해 연구되는 Information science 즉 정보과학의 시대로 접어들었다는 사실이었다.
이는 게놈프로젝트 이후 생물학의 트렌드를 보면 극명히 드러나는데, 이 후에 생물학의 세부 분야들은 Genome 처럼 총체라는 뜻을 더해주는 -ome이 붙는 High-Thoughput data science 로 이름이 붙여졌고 ( Exressome, Proteome, Interactome, Metabolom , etc ) , 뛰어난 생물학 연구 팀에는 의례히 뛰어난 Bioinformatics 인력 혹은 공동연구팀이 함께 연구를 이끌어 가고 있음을 확인할 수 있다. 다시 말하면, 이제 첨단 생물학을 연구하기 위해서는 bioinformatics 로 대변되는 정보 과학적 분석 능력이 필수가 되었다는 말이 된다.
최근에 게놈 분석 비용이 획기적으로 낮아지면서, 게놈의 대중화와 이와 연계된 산업적 폭발이 목전에 와 있는데, 여기서도 게놈의 분석/관리/서비스를 위한 Bioinformatics 인력이 주도적 역할을 하게될 터인데,
게놈프로젝트 이후 많은 대학에 학위 과정이 개설되어 많은 인력을 배출해낸 미국에 비해 한국은 그나마 교육과정을 개설했던 학교들에서 오히려 Bioinformatics 이외의 분야들로 타겟을 바꾸어 학과를 포지셔닝 하면서, 제대로된 Bioinformatics 교육 커리큘럼을 갖춘 학교가 전무한 실정이다. 사실 미국을 제외하면 Bioinformatics 학위 과정이 활성화된 나라가 거의 없긴 한데, 그래서 자연히 앞으로의 Bioinformatics 분야도 미국이 주도적으로 발전시켜 나갈 수밖에 없지 않나 생각한다.
여담으로 현재 국내에서 중급 Bioinformatics 인력 조차도 구하기 쉽지 않은데, 앞으로 꾀나 오랜 시간 이런 흐름이 지속되지 않을가 조심스레 예측해 본다.