꼭 참여 하실 분들은 contact@geference.com 메일로 등록해 주세요.
Mainly on Personal Genome Industry, Preventable Medicine, Mobile healthcare tech, and Bioinformatics.
Tuesday, May 28, 2013
Wednesday, April 10, 2013
노령화, 의료시스템의 위기, 정부의 역할 :: 영국의 국가 유전체 연구 전략을 통해 본 의료시스템과 국가 정책
영국의 국가 주도 대규모 Genome 프로젝트
영국은 프레데릭 생어에 의해 최초로 DNA sequencing이 탄생한 곳이고, 그 생어를 기려 웰컴트러스트의 기부로 설립한 Sanger institute 은 현재도 세계 최고의 유전체 연구소로 자리 메김하고 있다. 대규모 유전체 연구를 가능케 하는 Bioinformatics 도 EBI 와 Sanger 연구소를 통해 세계 최고 수준을 유지하며 유전체 연구를 위한 인프라도 부족함 없이 잘 구축되어 있다 할 수 있다.
하지만, 여느 field와 마찬가지로, 넓은 시장과 막강한 자금력을 바탕으로한 미국의 대학과 회사들이 유전체 학계와 시장을 주도하기 시작했고, 영국도 여느 나라와 마찬가지로 미국의 lead에 동참하는 top tier 중 한 곳으로 주저 앉은 느낌을 지울 수 없었던 것이 사실이다.
하지만 최근의 영국의 행보를 보면, 국가적으로 미래 유전체 market을 leading 하려는 강력한 의지가 보이는데, 그 근거는 최근 발표한 10만명 whole genome 프로젝트 그리고 50만명 genotyping 프로젝트.
노령화, 암울한 미래 의료 시스템
영국이 이런 엄청난 프로젝트를 진행하는 이유는 이 프로젝트의 주체 NHS 를 생각하면 쉽게 답이 나온다. NHS는 우리로 치면 의료보험건강공단 쯤 되는 곳인데, 이를 통한 영국의 의료시스템은 공짜다. 누구나 무상으로 병원을 이용할 수 있고, 약값은 어떤 약을 처방받든 일정 금액을 지불한다 . 이렇게 하면서도 사보험 중심 의료시스템의 미국이 2010년 기준 GDP 18%를 의료비로 소비하는데 반해, 영국은 그 절반 수준인 9% 정도만 의료비로 사용하는 효율적인 의료시스템을 운영하고 있다.
하지만, 고령화 추세에 발맞추어 의료비도 빠르게 상승하고 있다. 2000년대 초반 6%대에 머물던 것이, 2010년 기준 9%를 넘어섰고, 이런 추세가 이어지면 2020년 경에는 20%를 넘어서게 될 것으로 예상된다 ( 아래 그래프 참조, 고령화 추세는 developed country 들엔 공통적 고민거리고 한국도 마찬가지 추세가 확인된다. 남의 일이 아니다 ).
결국 모든 국가들이 장기적으로 급격히 늘어나는 의료비 문제에 봉착하게 되는 것이 현실이고, 현재의 의료 시스템은 어떻게든 혁신이 일어나야만 암울한 미래의 의료시스템의 붕괴를 피할 수 있는 상황이다.
유전체 의학 ( Genomic Medicine )과 의료 혁신
의료시스템의 혁신은 가장 상황이 심각하고 시급한 미국을 중심으로, 활발하게 다양한
대안 모델들이 시도 되고 있다. 원격 의료, 원격 처방, 전문 간호사 진료 ( 의사를 거치지 않고, diagnosis - prescription 이 명확한 질환들을 전문간호사의 관리하에 진료-처방 ) , Mobile 기술을 활용한 다양한 healthcare 등.
이러한 줄기들 중, 의료 혁신에 강력한 drive를 걸어줄 것으로 대두되는 것이 바로 개인유전체 혁명에 기반한 유전체 의학 ( Genomic Medicine )이다. 개개인의 유전체( Personal Genome ) 정보를 기반으로 개인의 healthcare 관리를 일상의 영역으로 끌어내려, 질병에 걸리기 이전에 미리 질병을 예방하는 예방의학( Preventible Medicine ) 을 가능케 하고, 암과 같은 치명적 질병을 개개인의 질병 발현을 일으킨 변이 유전자를 찾아내 개인화된 치료를 가능케 하고, 개인의 유전적 특성에 기초해 부작용을 일으키는 약물을 속아내거나, 안전한 수준의 약물 투여량 등을 판별해 내는 등의 일들이 개인의 유전체 정보를 의학에 이용할 때 가능해지는 일들이 되고, 국가 전체의 의료 시스템의 측면에서 보자면, 유전체 정보를 활용하여 효율적인 치료와 처방 및 예방이 가능해지면, 엄청난 의료비 지출 감소 효과를 기대할 수 있을 것으로 예상된다.
인간게놈프로젝트( Human Genome Project)에서 한 사람의 유전체 분석( DNA sequencing)에 3조원이 소모되었던 것이, 지금은 수백만원 수준으로 떨어졌고, 전체 유전체 중 유전자 영역인 Exome 부위의 DNA 만 읽어내는 Exome sequencing의 경우 소비자 가격이 이미 백만원 아래로 떨어진 상태다. MRI, CT 찍는 정도의 가격으로 전체 유전자 영역의 DNA sequencing이 가능하다는 말이고, 이는 곧, 지금 당장도 유전체 의학을 적용해 볼 수 있는 시대라는 것.
하지만, 아직 유전체 의학이 기대 만큼 제대로 환자의 치료에 적용되지는 못하는데, 이는 유전체와 질병 및 약물 등과의 연관성이 충분히 연구되지 않은 상태이기 때문이다.
10만명 Genome 프로젝트에 담긴 영국의 전략
앞서 언급한 영국 NHS 주도 10만명 Whole Genome 프로젝트는 유전체 의학의 실효성을 높일 유전체-질병,약물 연관성 정보의 부족을 타개하여, 유전체 의학의 실질적 이용을 가능케 하기 위해 디자인된 프로젝트다.
영국 국가 건강 서비스 NHS( National Health Service ) 주도로 암이나 희귀질병을 가진 영국인 10만명의 DNA sequence 30억개 전체 whole genome을 분석하는 인류 역사상 최대의 genome project 를 통해 질병과 유전체의 상관 관계에 대한 다양한 정보를 얻어내, 의료시스템에 활용하겠다는 전략. 영국 총리 데이빗 카메론은 "영국이 mainstream 의료 시스템에 유전체를 이용하는 최초의 국가가 될 것" 이라고 얘기하는데, 바로 이 프로젝트의 목적을 잘 보여주는 대목이다.
영국은 프레데릭 생어에 의해 최초로 DNA sequencing이 탄생한 곳이고, 그 생어를 기려 웰컴트러스트의 기부로 설립한 Sanger institute 은 현재도 세계 최고의 유전체 연구소로 자리 메김하고 있다. 대규모 유전체 연구를 가능케 하는 Bioinformatics 도 EBI 와 Sanger 연구소를 통해 세계 최고 수준을 유지하며 유전체 연구를 위한 인프라도 부족함 없이 잘 구축되어 있다 할 수 있다.
하지만, 여느 field와 마찬가지로, 넓은 시장과 막강한 자금력을 바탕으로한 미국의 대학과 회사들이 유전체 학계와 시장을 주도하기 시작했고, 영국도 여느 나라와 마찬가지로 미국의 lead에 동참하는 top tier 중 한 곳으로 주저 앉은 느낌을 지울 수 없었던 것이 사실이다.
하지만 최근의 영국의 행보를 보면, 국가적으로 미래 유전체 market을 leading 하려는 강력한 의지가 보이는데, 그 근거는 최근 발표한 10만명 whole genome 프로젝트 그리고 50만명 genotyping 프로젝트.
노령화, 암울한 미래 의료 시스템
영국이 이런 엄청난 프로젝트를 진행하는 이유는 이 프로젝트의 주체 NHS 를 생각하면 쉽게 답이 나온다. NHS는 우리로 치면 의료보험건강공단 쯤 되는 곳인데, 이를 통한 영국의 의료시스템은 공짜다. 누구나 무상으로 병원을 이용할 수 있고, 약값은 어떤 약을 처방받든 일정 금액을 지불한다 . 이렇게 하면서도 사보험 중심 의료시스템의 미국이 2010년 기준 GDP 18%를 의료비로 소비하는데 반해, 영국은 그 절반 수준인 9% 정도만 의료비로 사용하는 효율적인 의료시스템을 운영하고 있다.
하지만, 고령화 추세에 발맞추어 의료비도 빠르게 상승하고 있다. 2000년대 초반 6%대에 머물던 것이, 2010년 기준 9%를 넘어섰고, 이런 추세가 이어지면 2020년 경에는 20%를 넘어서게 될 것으로 예상된다 ( 아래 그래프 참조, 고령화 추세는 developed country 들엔 공통적 고민거리고 한국도 마찬가지 추세가 확인된다. 남의 일이 아니다 ).
 |
| 1995년 부터 2010년 까지 각 국가의 GDP 대비 의료비 지출을 표시한 그래프. X 축은 1인당 GDP, Y축이 GDP 대비 의료비 지출% |
GDP대비가 아닌, 정부 예산 대비 의료비 지출로 따지면, 영국은 이미 15% 이상을 의료비로 지출하고 있다( 아래 그래프 참조 , 미국은 20%를 이미 넘어섰고, 한국도 이미 12% 이상을 지출하고 있다 )
 |
| 2010년의 각 정부 지출 대비 의료비 지출을 표시한 그래프. |
결국 모든 국가들이 장기적으로 급격히 늘어나는 의료비 문제에 봉착하게 되는 것이 현실이고, 현재의 의료 시스템은 어떻게든 혁신이 일어나야만 암울한 미래의 의료시스템의 붕괴를 피할 수 있는 상황이다.
유전체 의학 ( Genomic Medicine )과 의료 혁신
의료시스템의 혁신은 가장 상황이 심각하고 시급한 미국을 중심으로, 활발하게 다양한
대안 모델들이 시도 되고 있다. 원격 의료, 원격 처방, 전문 간호사 진료 ( 의사를 거치지 않고, diagnosis - prescription 이 명확한 질환들을 전문간호사의 관리하에 진료-처방 ) , Mobile 기술을 활용한 다양한 healthcare 등.
이러한 줄기들 중, 의료 혁신에 강력한 drive를 걸어줄 것으로 대두되는 것이 바로 개인유전체 혁명에 기반한 유전체 의학 ( Genomic Medicine )이다. 개개인의 유전체( Personal Genome ) 정보를 기반으로 개인의 healthcare 관리를 일상의 영역으로 끌어내려, 질병에 걸리기 이전에 미리 질병을 예방하는 예방의학( Preventible Medicine ) 을 가능케 하고, 암과 같은 치명적 질병을 개개인의 질병 발현을 일으킨 변이 유전자를 찾아내 개인화된 치료를 가능케 하고, 개인의 유전적 특성에 기초해 부작용을 일으키는 약물을 속아내거나, 안전한 수준의 약물 투여량 등을 판별해 내는 등의 일들이 개인의 유전체 정보를 의학에 이용할 때 가능해지는 일들이 되고, 국가 전체의 의료 시스템의 측면에서 보자면, 유전체 정보를 활용하여 효율적인 치료와 처방 및 예방이 가능해지면, 엄청난 의료비 지출 감소 효과를 기대할 수 있을 것으로 예상된다.
인간게놈프로젝트( Human Genome Project)에서 한 사람의 유전체 분석( DNA sequencing)에 3조원이 소모되었던 것이, 지금은 수백만원 수준으로 떨어졌고, 전체 유전체 중 유전자 영역인 Exome 부위의 DNA 만 읽어내는 Exome sequencing의 경우 소비자 가격이 이미 백만원 아래로 떨어진 상태다. MRI, CT 찍는 정도의 가격으로 전체 유전자 영역의 DNA sequencing이 가능하다는 말이고, 이는 곧, 지금 당장도 유전체 의학을 적용해 볼 수 있는 시대라는 것.
하지만, 아직 유전체 의학이 기대 만큼 제대로 환자의 치료에 적용되지는 못하는데, 이는 유전체와 질병 및 약물 등과의 연관성이 충분히 연구되지 않은 상태이기 때문이다.
10만명 Genome 프로젝트에 담긴 영국의 전략
앞서 언급한 영국 NHS 주도 10만명 Whole Genome 프로젝트는 유전체 의학의 실효성을 높일 유전체-질병,약물 연관성 정보의 부족을 타개하여, 유전체 의학의 실질적 이용을 가능케 하기 위해 디자인된 프로젝트다.
영국 국가 건강 서비스 NHS( National Health Service ) 주도로 암이나 희귀질병을 가진 영국인 10만명의 DNA sequence 30억개 전체 whole genome을 분석하는 인류 역사상 최대의 genome project 를 통해 질병과 유전체의 상관 관계에 대한 다양한 정보를 얻어내, 의료시스템에 활용하겠다는 전략. 영국 총리 데이빗 카메론은 "영국이 mainstream 의료 시스템에 유전체를 이용하는 최초의 국가가 될 것" 이라고 얘기하는데, 바로 이 프로젝트의 목적을 잘 보여주는 대목이다.
이 프로젝트는 구체적으로 2014년 4월까지 10만명의 whole genome sequencing을 수행할 기관을 선정하고, 프로젝트를 진행할 계획이라고 하는데, 프로젝트 에는
- 유전체 의학, 약물유전체학을 연구할 전문 인력 교육
- 대규모 유전체 정보를 관리할 computer system 구축
에 대한 계획도 수립되어 있다고 한다.
이 프로젝트가 성공적으로 완수가 되면 영국은
- 세계 최대의 유전체 의학 정보 knowledgebase 구축( 이 자체가 엄청난 자산)
- 유전체 의학의 NHS 이용으로 국가적 의료비 절감
- 프로젝트를 통해 대규모 유전체 정보 분석 전문 인력 대규모로 양성
- 대규모 유전체 정보 flow 컨트롤 가능한 computer system 구축 ( 현재 IT의 최대 화두 중 하나인 Cloud 컴퓨팅 환경 하에서의 Genome data 활용에 필요한 기반 기술의 개발이 기대됨 )
등의 효과를 기대할 수 있는데, next paradigm인 Genome 산업에 매우 유리한 위치를 선점하게 되는 것.
이런 대단한 프로젝트에 드는 돈은 얼마나 될까? 겨우 $160 million 이다. 겨우 우리돈 2천억 정도. 4대강에 수십조 쏟은 우리의 경험을 생각해 보면, 겨우 2천억 정도 푼돈을 쏟아 Genome 대국이 되는 영국의 정책 입안자들이 그저 부러울 따름이다.
50만명 Genotyping 프로젝트 :: 또다른 영국의 국가 주도 유전체 프로젝트
10만명 Genome 프로젝트 이외에 영국은 UK Biobank 주관의 50만명 Genotyping 프로젝트도 시작한다.
이 프로젝트는 40세에서 69세 사이의 영국인인 50만명의 blood, urine, saliva 샘플을 Affymetrix Genotyping chip을 이용해 분석하여, 다양한 질병과 유전체의 상관관계를 밝혀내는 것이 목적이다. 10만명 프로젝트와 차이는 이것이 추적조사연구( longitudinal study) 라는 점.
참여자의 medical history, lifestyle( diet, 다양한 설문, body scan, 그외 accelerometer를 이용한 activity tracking 등도 고려) 그리고 다양한 테스트 결과들( fMIR, CT scan, X-ray 등의 정보의 포함 까지 계획)이 유전체 정보와 함께 분석되어 구체적인 유전체와 질병 발병의 상관 관계를 밝혀낼 수 있게 된다.
질병의 발현은 개인의 유전체 타입으로만 결정되는 것이 아니라, 다양한 생활 습관, 식습관, 운동, 환경적 요인등 다양한 생활-환경 요소가 함께 결합되어 결정이 된다. 이 프로젝트에서는 개인의 유전체 뿐만 아니라 다양한 개인의 생활-환경 요소 정보를 함께 축적하여 질병의 발현의 종합적 분석을 하려는 목적을 담고 있다.
이런 시도는 인류 역사상 최초로 시도되는 가장 구체적인 질병 연구라 할 수 있고, 여기서 쏟아지는 질병 정보는 인류가 지금까지 축적한 질병의 발현 정보를 기하 급수적으로 확장시킬 것이라 예상한다.
이 프로젝트 또한 $160million 정도, 우리돈 2천억 정도만 소모된다고 한다.
현재 미국과 중국이 이와 유사한 프로젝트를 계획하고 있다고 하는데, 미국은 이 프로젝트에 $2 billion 우리돈 2조 이상을 투입할 것이라 한다. ( 얼마나 대규모로 진행할려고 2조 이상을 ... )
이 두 프로젝트 도합 우리돈 4천억 밖에 안 든다. 도로명 주소 까는데 4천억 이상의 예산이 소모되었다고 한다. 통크게 1조 정도 써서 50만명 whole genome 기반 10년 longitudinal 질병 프로젝트 하면, 한국은 유전체 연구, 산업 Top tier 국가로 올라선다.
앞서나가는 국가들, 그리고 한국
한국은 지금까지 whole genome sequencing 이 가장 많이 수행된 국가 중 한곳으로, 다양한 genome 관련 연구 프로젝트가 진행되고 있긴 하다. BT가 성장 동력이라는 것도 잘 인식하고 있고, 이를 위해 다양한 정책과 산업적 움직임이 감지되고 있다. 하지만, Genome 산업과 그 정책에 대해서만 따져보면, 의료시스템과 Genome 기술과 산업을 한데 묶어, 경쟁력 있는 미래를 위한 정책이나 투자는 전혀 진행되고 있지 못하다고 생각한다.
유전체 산업에 대해서만 보자면, 다양한 idea들이 시장에 선보이고 자연스럽게 시장에 의해 성공과 실패가 반복되며 경쟁력 있는 유전체 산업과 제품군이 생겨나는 창의적인 순환이 일어나야 하지만, 생명의료윤리법에 의해 이러한 시도는 완전히 불가능하게 막혀 있다.
미국이 모두 정답은 아니지만, GINA법( 개인의 유전적 특질로 인해 차별받지 않을 권리)이나, 소비자유전학에 대한 관리규정 등 사회,윤리적 문제가 될 만한 부분들을 법적 장치나 관리 기관의 관리 하에 허용하면서, 유전체 산업의 다양한 창의적 시도들이 가능한 환경은 정말 부럽기 그지 없다.
유전체 산업에서는 앞으로 분명히 구글, 애플을 능가하는 회사가 출현할 것인데, 어떤 비즈니스 모델이 이런 회사로 이어질지는 아무도 모른다. 자연히 다양한 시도를 하는 중에 이런 회사가 툭 튀어 나오게 된다. 실리콘밸리의 성공을 굳이 언급할 필요는 없을터.
IT에서 실리콘 밸리가 성공했는데, BT 유전체 산업에서도 지금 이런 일이 반복되고 있다. 인간 유전체 산업은 사회, 윤리적 문제가 여타 산업과 비교할 수 없을 만큼 파급력이 강할 수밖에 없다. 하지만, 무엇이 문제가 될 것인지, 이런 문제를 어떻게 보호할지는 적절한 법적인 울타리를 만들어 컨트롤 하는 것이 가능하다. 당장 문제가 되는 부분들을 법제화하고, 새로운 문제는 그 때 그때 만들어 나가면 된다.
그런 울타리를 치고, 창의적 아이디어들이 시장에 살아 숨쉴 수 있도록 만들어야 한다. 선진국들이 모든 문제를 해결해 놓고 난 후에, 이를 그저 답습하는 식의 전략은 이제 그만 버려야 한다. 적극적으로, 주도적으로 목표를 세우고 문제를 해결해 나가는 자세를 국가가 견지 하지 않는 한, next big thing BT 산업에서 한국이 선도국가로 발돋움하는 일은 없을거라 '확신'한다.
Monday, April 8, 2013
생물정보학은 BT와 IT의 결합이 아니다.
Bioinformatics를 BT 전문가 분들도 단순히 BT와 IT의 결합이라고 생각하는 경우가 많음을 지난주 GMIG 미팅에서 느꼈다. 단순히 BT 하는 사람과 IT 하는 사람들이 같이 모여서 열심히 일하면 잘 되는 문제로 생각하는 경향이 있는데, 절대 그렇게 해서 bioinformatics가 제대로 안 된다.
달리기 잘하는 사람과 테니스 잘 치는 사람 둘을 모으면 좋은 야구팀이 될까? 야구는 야구를 전문적으로 연습한 사람이 테니스 세계 1위 보다, 100m 기록 보유자 우사인 볼트보다 훨씬 잘 한다.
바이오인포매틱스도 마찬가지. 세부적으로 도구를 나눠 보자면, BT도 알아야 하고 IT 도 알아야 하니, 이 둘을 잘 하는 사람을 모아 놓으면 잘 할 것 같지만, 실상은 전혀 아니다. BT하는 사람은 IT의 철학과 방법론을 모르고, IT는 다루는 데이터의 본질을 알지 못한다. 이렇게 교집합 부분이 텅 비어 있는 상태로 올바른 합집합이 만들어 지지 않는다.
바이오인포는 바이오인포를 전문적으로 학습하고 시간을 투자하여, 바이오 데이터를 IT 적 시각으로 보고, 통계적으로 바라볼 수 있는 안목이 있어야 비로소 제대로 된 일을 수행할 수 있다.
경험상 어느 한쪽에 전문성을 가진 사람이 바이오인포로 와서 반대쪽에 익숙해 지는데는 적어도 학위 과정 정도의 시간이 필요하다. 굳이 더 따지자면 BT-> IT가, IT->BT로 가는 경우 보다 더 짧은 시간 안에 좋은 성과를 낸다.
얼핏 생각하면 생물학이 공부하기 더 쉬우니 IT->BT 로 가는 것이 더 빠르지 않을까 생각할 수 있는데, 바이오인포 즉 생물정보학 = 생물학 이다. 바이오인포는 결국 생물학 문제를 풀어야 의미가 있는 일이다. 자연히 생물학적 지식에 기반한 문제 인식을 가진 생물학 기반의 생물정보학자가 반대쪽의 경우보다 '본질'을 더 잘 이해하고 접근하는 경우가 많다.
IT에서 온 사람들이 이런 노력을 등한시 하는 경우, 그저 algorithm 최적화 문제에 집착하고 시간을 낭비하는 경우가 많다. 하지만, 그건 생물정보학이 아니라, 그냥 알고리즘 문제 해결일 뿐이고, 의미 있는 일도 아니다.
반대로 생물학에서 넘어온 사람들은 기초적인 프로그래밍 능력만 갖추어도 의미 있는 생물학 문제에 의미 있는 수준의 결과를 내는 것이 가능하다. fancy한 알고리즘이나 알고리즘의 효율성은 전혀 문제가 되지 않기 때문이다. ( 하지만, 제대로 된 알고리즘 설계를 하지 못한다면, 풀지 못하는 생물정보학의 문제가 매우 많은 것이 현실 )
생물정보학은 아주 전도 유망한 학문이다. 왜? 생물정보학은 현대 사회가 요구하는 데이터 분석 능력(big data 의 통계적 분석 능력), IT 이해( 프로그래밍 능력, 웹, 클라우드를 컨트롤 하는 능력), 그리고 next paradigm 인 유전체/의학을 이해하는 사람을 길러내는 학문이기 때문이다.
현재 미국에서 생물정보학자가 평균적으로 생물학자보다 연봉이 50% 높다. ( 스탠포드의 생물정보학자들이 인터뷰에서 2-3배 라고 했는데, 평균적으로 따지면 그 보다는 낮다. 하지만, 실질 체감 연봉은 그 정도 차이라는 것 ) 앞으로 이런 갭은 더욱 커질 거라 보는데, 이는 생물정보학자가 선택할 수 있는 도메인이 늘어날 것이기 때문.
Sunday, April 7, 2013
지금 BT에 필요한 IT 기술이란?
아래 내용은 GMIG( Genomic Medicine Interest Group ) 2회 모임 후 관련 내용을 멤버들과 share한 내용으로, 내용의 변경 없이 그대로 여기에 남깁니다.
-------------------------------------------------------------------------------------
2회 모임의 내용 중에 제(금창원)가 지적하고 싶었던 내용이 하나더 있는데, 그것은 바로 IT에 대한 '환상'의 경계 입니다. 박재범 대표님의 발표를 보면서 그런 생각이 들었는데, 재범 대표님의 발표에서 중요하게 받아들여야 할 것은 IT의 영역 중 '소비자 중심의 UX / UI 와 관련한 부분'들로 BT에 적용되지 않았던 '일반 대중'을 대상으로 접근성이 높고, 지속가능한 유용성을 꾸준히 제공하는 IT application이 필요하다는 점 이라고 봅니다.
이런 부분은 인류 역사상 BT에 거의 접목이 된 적이 없는 부분에 가깝다 봅니다. 근접하는 성공으로 한국인 정세주 대표가 뉴욕에서 창업한 Noom의 다이어트앱 정도를 들 수 있을 것 같습니다. ( 최초엔 running trajectory tracing과 칼로리 소모량 기록용 개인 data log 앱이었으나, 발전하여 지금은 식습관 등이 포함되어 살빼는 목적을 실현시켜주는 앱이 되었고 전세계 user가 1500만명이 넘습니다. )
이런 식의 접근이 Genetics에도 시도가 되어 나가면 엄청난 혁신이 있을거라는 것을 GMIG 참여자 분들이라면 충분히 이해하시리라 생각 합니다.
하지만, Genomics의 연구 관점에서 보자면 이미 최첨단의 IT 기술들이 충분히 적용되고 있고, 경우에 따라서는 BT inspired IT 기술이 IT의 최첨단 기술이 되고 있습니다.
당장 현재 IT 최대 화도 cloud 컴퓨팅만 해도 유전체 데이터 분석이 major application domain으로 활발하게 연구 개발이 되고 있죠( KT에서 GenomeCloud를 베타 테스트 중입니다)
재범 대표님의 발표에서 구글의 Pagerank 알고리즘이 적용된 Gene priotization 연구를 언급하셨는데, 이런 연구는 gene expression 연구 초창기인 2000년대 초반 부터 minor 저널에 심심하면 한번씩 출판되던 내용입니다. 그냥 구글 서치해보면 2005년 논문이 상위에 뜨네요. 왜 이런 논문이 minor 저널에 출판되냐 하면, 당연히 value가 낮기 때문입니다.
즉, 이런 알고리즘을 적용하는 것은 fancy한 접근일 수는 있지만, '문제 해결'은 되지 않기 때문입니다. 기존의 방법론 보다 더 나은 해결책을 제시해 주지도 못하는데 그 이유는 간략하게 생물체의 variation은 엄청나기 때문이라는 것 정도만 여기에 언급해 둡니다. Gene expression과 관련해선 FDA의 MAQC( Microarray Quality Control ) 컨소시움의 연구를 통해 이런 점들이 이미 충분히 지적이 되었습니다.
(MAQC 내용을 정리한 제 블로그 글http://goldbio.blogspot.kr/2011/03/20.html )
MAQC는 당시 가능한 모든 통계적 분석법과 machine learning 알고리즘을 gene expression 분석에 적용해 보고, 가장 consistent하고 재현성 높은 분석 기준을 찾는 것을 목적으로 미국의 51개 major 유전체 연구기관이 참여한 대규모 study 였습니다.
기대는 뭔가 첨단의 hign-end 방법론이 best 일거라 기대했지만, 그 예상은 처참히 무너집니다. 최고는 초창기 부터 일반적으로 써왔던 fold change ( 그냥 단순히 case-control의 비율) 과 가장 간단한 t-test를 적절히 혼용하는 것이었죠.
재범 대표 님이 발표한 내용 중 network analysis 와 관련된 내용도 있었는데, network analysis 는 네트웍 분석이 바라바시에 의해 중요하게 부각된 초창기 부터 BT가 major application domain 으로 2000년대 초반부터 BT 특히나 bioinformatics에서는 일상적으로 다루어져 오고 있습니다. 바라바시가 쓴 그 유명한 저서 'Linked'에도 현재 테라젠의 박종화 연구소장님의 단백질 구조 도메인의 네트웍 연구가 소개되어 있을 정도 입니다.
심지어 network visualization 소프트웨어 중 최고로 인정되는 것이 바로 요즘 생물학 연구실에서 한명 쯤은 쓰고 있는 cytoscape라는 오픈 소스 소프트웨어 입니다. ( 이는 리로이 후드가 이끄는 ISB 에서 개발한 소프트웨어 )
사실 BT의 연구 측면에서만 보자면 IT 적 시각을 가진 사람들( bioinformatician )이 해야하는 역할은 새로운 시각으로 문제 해결의 새로운 가능성을 여는 일이라 봅니다. ( 이에 대해서도 일전에 정리해 둔 글을 참고로 링크 합니다http://goldbio.blogspot.kr/2011/02/blog-post.html )
Broad나 Sanger 연구소가 유전체 연구의 최고로 인정받는 이면에는 바로 이런 역할을 하는 사람들이 directing board로 참여하면서 연구의 혁신을 이끌기 때문입니다. ( sanger의 리차드 더빈, 팀 허바드 broad의 토드 골럽 등) 국외 뿐만 아니라, 국내에서도 유전체 연구를 선도하는 테라젠이나 서정선 교수님이 이끄는 서울대유전체연소(GMI) 등도 훌륭한 bioinformatician( 혹은 cross-functional researcher ) 들의 존재 때문에 선구적인 연구들을 진행할 수 있다고 봅니다.
글이 길어졌는데, 정리해 보면, IT의 UX/UI 기술들이 접목이 되어야 결국 진정한 personal genomics로 갈 수 있고, 대중화될 수 있다는 점. 하지만 연구자의 입장에서는 이미 충분히 첨단의 IT 기술이 적용되고 있고, 필요한 내용들은 대부분 쉽게 사용할 수 있는 application들이 존재하니, 활용을 하고, IT 기술에 대한 환상을 가질 필요는 없다는 점, 그리고 fancy한 기술 보다는 어떤 문제를 해결하는데 새로운, 참신한 idea를 간단한 IT 기법들로, crude한 상태라도 적용해보는 것이 중요할 수 있다는 아이디어들을 나누고 싶었습니다
-------------------------------------------------------------------------------------
2회 모임의 내용 중에 제(금창원)가 지적하고 싶었던 내용이 하나더 있는데, 그것은 바로 IT에 대한 '환상'의 경계 입니다. 박재범 대표님의 발표를 보면서 그런 생각이 들었는데, 재범 대표님의 발표에서 중요하게 받아들여야 할 것은 IT의 영역 중 '소비자 중심의 UX / UI 와 관련한 부분'들로 BT에 적용되지 않았던 '일반 대중'을 대상으로 접근성이 높고, 지속가능한 유용성을 꾸준히 제공하는 IT application이 필요하다는 점 이라고 봅니다.
이런 부분은 인류 역사상 BT에 거의 접목이 된 적이 없는 부분에 가깝다 봅니다. 근접하는 성공으로 한국인 정세주 대표가 뉴욕에서 창업한 Noom의 다이어트앱 정도를 들 수 있을 것 같습니다. ( 최초엔 running trajectory tracing과 칼로리 소모량 기록용 개인 data log 앱이었으나, 발전하여 지금은 식습관 등이 포함되어 살빼는 목적을 실현시켜주는 앱이 되었고 전세계 user가 1500만명이 넘습니다. )
이런 식의 접근이 Genetics에도 시도가 되어 나가면 엄청난 혁신이 있을거라는 것을 GMIG 참여자 분들이라면 충분히 이해하시리라 생각 합니다.
하지만, Genomics의 연구 관점에서 보자면 이미 최첨단의 IT 기술들이 충분히 적용되고 있고, 경우에 따라서는 BT inspired IT 기술이 IT의 최첨단 기술이 되고 있습니다.
당장 현재 IT 최대 화도 cloud 컴퓨팅만 해도 유전체 데이터 분석이 major application domain으로 활발하게 연구 개발이 되고 있죠( KT에서 GenomeCloud를 베타 테스트 중입니다)
재범 대표님의 발표에서 구글의 Pagerank 알고리즘이 적용된 Gene priotization 연구를 언급하셨는데, 이런 연구는 gene expression 연구 초창기인 2000년대 초반 부터 minor 저널에 심심하면 한번씩 출판되던 내용입니다. 그냥 구글 서치해보면 2005년 논문이 상위에 뜨네요. 왜 이런 논문이 minor 저널에 출판되냐 하면, 당연히 value가 낮기 때문입니다.
즉, 이런 알고리즘을 적용하는 것은 fancy한 접근일 수는 있지만, '문제 해결'은 되지 않기 때문입니다. 기존의 방법론 보다 더 나은 해결책을 제시해 주지도 못하는데 그 이유는 간략하게 생물체의 variation은 엄청나기 때문이라는 것 정도만 여기에 언급해 둡니다. Gene expression과 관련해선 FDA의 MAQC( Microarray Quality Control ) 컨소시움의 연구를 통해 이런 점들이 이미 충분히 지적이 되었습니다.
(MAQC 내용을 정리한 제 블로그 글http://goldbio.blogspot.kr/2011/03/20.html )
MAQC는 당시 가능한 모든 통계적 분석법과 machine learning 알고리즘을 gene expression 분석에 적용해 보고, 가장 consistent하고 재현성 높은 분석 기준을 찾는 것을 목적으로 미국의 51개 major 유전체 연구기관이 참여한 대규모 study 였습니다.
기대는 뭔가 첨단의 hign-end 방법론이 best 일거라 기대했지만, 그 예상은 처참히 무너집니다. 최고는 초창기 부터 일반적으로 써왔던 fold change ( 그냥 단순히 case-control의 비율) 과 가장 간단한 t-test를 적절히 혼용하는 것이었죠.
재범 대표 님이 발표한 내용 중 network analysis 와 관련된 내용도 있었는데, network analysis 는 네트웍 분석이 바라바시에 의해 중요하게 부각된 초창기 부터 BT가 major application domain 으로 2000년대 초반부터 BT 특히나 bioinformatics에서는 일상적으로 다루어져 오고 있습니다. 바라바시가 쓴 그 유명한 저서 'Linked'에도 현재 테라젠의 박종화 연구소장님의 단백질 구조 도메인의 네트웍 연구가 소개되어 있을 정도 입니다.
심지어 network visualization 소프트웨어 중 최고로 인정되는 것이 바로 요즘 생물학 연구실에서 한명 쯤은 쓰고 있는 cytoscape라는 오픈 소스 소프트웨어 입니다. ( 이는 리로이 후드가 이끄는 ISB 에서 개발한 소프트웨어 )
사실 BT의 연구 측면에서만 보자면 IT 적 시각을 가진 사람들( bioinformatician )이 해야하는 역할은 새로운 시각으로 문제 해결의 새로운 가능성을 여는 일이라 봅니다. ( 이에 대해서도 일전에 정리해 둔 글을 참고로 링크 합니다http://goldbio.blogspot.kr/2011/02/blog-post.html )
Broad나 Sanger 연구소가 유전체 연구의 최고로 인정받는 이면에는 바로 이런 역할을 하는 사람들이 directing board로 참여하면서 연구의 혁신을 이끌기 때문입니다. ( sanger의 리차드 더빈, 팀 허바드 broad의 토드 골럽 등) 국외 뿐만 아니라, 국내에서도 유전체 연구를 선도하는 테라젠이나 서정선 교수님이 이끄는 서울대유전체연소(GMI) 등도 훌륭한 bioinformatician( 혹은 cross-functional researcher ) 들의 존재 때문에 선구적인 연구들을 진행할 수 있다고 봅니다.
글이 길어졌는데, 정리해 보면, IT의 UX/UI 기술들이 접목이 되어야 결국 진정한 personal genomics로 갈 수 있고, 대중화될 수 있다는 점. 하지만 연구자의 입장에서는 이미 충분히 첨단의 IT 기술이 적용되고 있고, 필요한 내용들은 대부분 쉽게 사용할 수 있는 application들이 존재하니, 활용을 하고, IT 기술에 대한 환상을 가질 필요는 없다는 점, 그리고 fancy한 기술 보다는 어떤 문제를 해결하는데 새로운, 참신한 idea를 간단한 IT 기법들로, crude한 상태라도 적용해보는 것이 중요할 수 있다는 아이디어들을 나누고 싶었습니다
Friday, February 1, 2013
소비자 유전학( DTC: Direct To Consumer ) pioneer 회사들의 현재
소비자 유전학, DTC(Direct To Consumer ) genetics, 회사라고 불릴 수 있는 곳의 효시는 Google의 co-founder 세르게이 브린의 와이프 앤 워치키가 설립한 23andMe 라고 할 수 있다. 좀 더 엄밀히 따지면, deCODE genetics가 몇일 앞서지만, DTC 서비스라는 비즈니스 모델을 세상에 널리 알리며 소비자유전학이란 말을 만들어 내게 된 공은 역시 23andMe 로 돌리는 게 맞다.
이 두 회사의 설립 후, 암 전문의로 LA 헐리웃에서 활동하면서 이미 유명세를 떨치던 USC 의대 교수 데이빗 아구스가 디트리히 스테판과 함께 설립한 Navigenics가 23andme와 deCODE genetics의 뒤를 이어 세번째로 DTC 회사 리스트에 이름을 올렸다. ( 데이빗 아구스는 이 후, 스티브 잡스의 췌장암을 Genome 분석으로 치료하려는 시도를 하게 되면서, 잡스의 주치의 팀에도 합류하게 된다 )
전자공학도 이자 4개의 벤처기업을 런칭한 경험이 있던 제임스 플랜트는 이후 4번째 DTC 회사인 Pathway Genomics 를 설립한다.
23andme : 설립 2006년 4월, 2007년 11월 DTC 서비스 런칭
deCODE genetics : 설립 1996년 2007년 11월 DTC 서비스 런칭
Navigenics : 설립 2007년 11월, 2008년 4월 DTC 런칭
Pathway genomics : 설립 및 DTC 런칭 2008년
23andme 는 아무래도 Google의 후광 효과와 가장 싼 DTC 서비스를 내세워, 현재까지 가장 많은 고객을 유치했고( 2012년말 18만명), 최근에는 또다시5천억 5백억 이상의 투자를 받으며 서비스 가격을 $99까지 낮추어 100만 고객 유치를 기치로 내걸고 박차를 가하고 있다.
사라진 두 pioneer
deCODE genetics의 DTC 서비스였던 deCODEme 는 크게 각광 받지 못하였고, 최근 다국적 제약사 Amgen에 4천억 상당에 인수 되었다. Amgen의 deCODE 인수는 deCODE가 쌓아둔 Genome knowledge DB 및 분석 자원(인력과 방법론)을 활용한 신약 개발 과정의 혁신을 위해서라는 분석이 지배적이다. Navigenics 역시 크게 각광 받지 못하다가 최근 CLIA-lab 확보를 위해 노력하던 Life Technologies 에 500억 상당에 인수 되었다.
두 회사를 인수한 Amgen과 Life tech 모두 DTC 서비스의 중단을 선언했다. 엄밀하게 Navigenics 서비스는 이미 중단되었고, deCODE의 서비스인 deCODEme 는 중단할 예정이라고 발표가 난 상태다.
Pathway genomics는 최근 Blue shield of california 라는 캘리포니아 사보험 사와 계약하여 새로운 판로를 뚫었는데, 이 전 부터 다이어트, 올림픽 운동 선수 건강 가이드라인 등에 이용 등 마케팅을 꾸준히 하고, 해외 시장을 적극적으로 개척하는 등의 노력을 하며 비즈니스 안정화에 많은 노력을 기울이고 있는 것으로 보인다.
이 두 회사의 설립 후, 암 전문의로 LA 헐리웃에서 활동하면서 이미 유명세를 떨치던 USC 의대 교수 데이빗 아구스가 디트리히 스테판과 함께 설립한 Navigenics가 23andme와 deCODE genetics의 뒤를 이어 세번째로 DTC 회사 리스트에 이름을 올렸다. ( 데이빗 아구스는 이 후, 스티브 잡스의 췌장암을 Genome 분석으로 치료하려는 시도를 하게 되면서, 잡스의 주치의 팀에도 합류하게 된다 )
전자공학도 이자 4개의 벤처기업을 런칭한 경험이 있던 제임스 플랜트는 이후 4번째 DTC 회사인 Pathway Genomics 를 설립한다.
23andme : 설립 2006년 4월, 2007년 11월 DTC 서비스 런칭
deCODE genetics : 설립 1996년 2007년 11월 DTC 서비스 런칭
Navigenics : 설립 2007년 11월, 2008년 4월 DTC 런칭
Pathway genomics : 설립 및 DTC 런칭 2008년
23andme 는 아무래도 Google의 후광 효과와 가장 싼 DTC 서비스를 내세워, 현재까지 가장 많은 고객을 유치했고( 2012년말 18만명), 최근에는 또다시
사라진 두 pioneer
deCODE genetics의 DTC 서비스였던 deCODEme 는 크게 각광 받지 못하였고, 최근 다국적 제약사 Amgen에 4천억 상당에 인수 되었다. Amgen의 deCODE 인수는 deCODE가 쌓아둔 Genome knowledge DB 및 분석 자원(인력과 방법론)을 활용한 신약 개발 과정의 혁신을 위해서라는 분석이 지배적이다. Navigenics 역시 크게 각광 받지 못하다가 최근 CLIA-lab 확보를 위해 노력하던 Life Technologies 에 500억 상당에 인수 되었다.
두 회사를 인수한 Amgen과 Life tech 모두 DTC 서비스의 중단을 선언했다. 엄밀하게 Navigenics 서비스는 이미 중단되었고, deCODE의 서비스인 deCODEme 는 중단할 예정이라고 발표가 난 상태다.
Pathway genomics는 최근 Blue shield of california 라는 캘리포니아 사보험 사와 계약하여 새로운 판로를 뚫었는데, 이 전 부터 다이어트, 올림픽 운동 선수 건강 가이드라인 등에 이용 등 마케팅을 꾸준히 하고, 해외 시장을 적극적으로 개척하는 등의 노력을 하며 비즈니스 안정화에 많은 노력을 기울이고 있는 것으로 보인다.
Saturday, January 19, 2013
개인 게놈의 의학적 이용에서 제기되는 문제점과 미래
어제 아산병원 이종극 박사님의 Lead로 closed meeting으로 개최되었던 'Genomic Medicine Interest Group Meeting' 은 다양한 전공의 과목 의사분들에 의해 실제로 환자 치료나 연구에서 Genome 을 이용한 사례들이 넘쳐나는 흥미롭고 유익한 모임이었다.
모임의 말미에 아산병원 신수용 선생님이 개인 게놈 정보를 실제 병원에서 의학적으로 이용함에 있어 해결해야할 굉장히 중요한 문제점들을 제시해 주셨고, 이에 대해 다양한 시각의 의견들이 제시가 되었다. 흥미로운 주제이기도 하고, 또 앞으로 Genome의 산업적 이용에 있어 매우 중요한 사안들이기에, 이렇게 글로 정리하여 많은 사람들과 나누는 것이 의미 있을 거라는 판단에 정리해 본다.
( 기억력의 한계로 제대로 정리되지 않은 부분이 포함되었을 수 있고 상당 부분 논의된 내용이 빠져 있고, 개인적으로 인상 깊었던 두가지 사안에 대해서만 정리를 했습니다. 혹, 잘못된 부분/추가/수정 해야할 부분이 있으면 지적해 주시면 감사하겠습니다 )
1. 개인 게놈 raw data 의 저장 문제
문제 개괄
현재 NGS machine에서 한 사람의 whole human genome을 30x 로 읽으면 fastq raw data size는 180GB(3GB*30*2 -for fastq quality score space- )가 넘어가는 Big Data가 생성되고, 이를 기반으로 많은 환자들의 치료에 이용하는 병원과 같은 곳의 Genome center는 심각한 data storage 문제가 발생한다. 흔히 이에 대한 해결책으로 얘기되고 있는 Cloud 시스템은 a) 이용 요금이 생각보다 싸지 않고, b) 한 사람의 raw data 전송에 수일이 소요되는 전송 속도의 문제 등으로 인하여 적절한 해결책이 되지 못할 공산이 크다.
이종극 박사님 의견
병원에서 raw data를 계속 저장하고 있을 필요가 없다. 데이터 분석에 이용되는 환경( reference genome 과 분석에 이용되는 DB 들의 update )의 변화로 설령 추후에 분석 내용이 변경될 수 있다고 하더라도, 병원에서 genome data의 효용은 환자를 치료하는 시점( at the point of care )에 국한되고, 이 시점이 지난 시점은 이미 데이터 효용이 사라진 후기 때문에 raw data를 가지고 있을 필요가 없다.
최형진 선생님( 충북대 내분비내과 ) 의견
현장의 의사는 현실적으로 개개의 환자를 진료하는데 많은 시간을 쏟을 수 없다. 의사에게 전달되는 Genome 분석 결과는 한두페이지 짜리 최종 분석 보고서면 충분하다. 또한 현재 환자 치료에 이 분석 내용을 활용하였다면, 추후 다시 이 환자의 genome raw data를 재분석해 활용해 얻는 이익은 미미할 것이고, 현실적으로도 이런 재분석이 필요한 경우는 흔하지 않을 것이다.
결론적으로 raw data 저장에 여러가지 현실적 제약이 있다면, 병원이 raw data를 계속 가지고 있을 필요는 없다고 본다.
개인적 의견
Genome raw data storage는 기술의 발전이 자연스럽게 해결해 줄 것이라고 생각한다. 사실 현재의 raw data 사이즈가 큰 이유는 NGS 기계들의 떨어지는 성능 탓이다. DNA 30억개 문자로 3G면 되는 개인 게놈을 왜 30X 즉 30배로 읽을까? a) NGS 기계들이 200 base 단위 정도의 짧은 조각으로 게놈을 읽고, b) 그 마저도 1% 정도의 에러가 섞이기 때문이다. 그래서 전체 게놈의 30배 정도로 200bp 길이의 짧은 조각 DNA read 들을 생산해 '퍼즐 맞추기' 과정을 통해 문제 a 를 해결하고, 같은 부분에 읽힌 30개의 DNA를 확인해 b 문제를 해결하는 형태로 현재의 NGS 기계들이 작동하고 있기 때문이다.
현재 내부적으로 fine-tuning 과정을 거치고 있는 Oxford nanopore 의 4세대 DNA sequencer 가 그들의 주장대로 기계의 성능을 갖춰 출시된다면, 개인 게놈 raw data 사이즈는 혁신적으로 작아지게 된다. Oxford 의 read 길이는 평균 1만 base ( 2012년 초 기준) , error rate 4%( 역시 2012년 초 기준) 다. Oxford는 2012년말 보도 자료에서 error rate 이 1% 이내가 되기 전에는 시장에 출시하지 않겠다고 발표했다. 즉, 시장에 나오는 Oxford 제품은 평균 read 길이 최소 1만에 1% 이하의 error rate의 read를 생산하는 획기적인 DNA sequencer 가 된다.
Oxford 이후에도 Sequencer 성능은 꾸준히 개선될 것이고, 일정 시점에선 whole genome을 그냥 한번 읽어내면 되는 Sequencer 완성형 제품이 나오게 될 것이라고 생각한다. 그렇게 되면 그냥 개인 게놈은 3G 가 된다. 3G 라면 raw data 저장 문제도 그냥 해결이 된다. 현재 우리가 공짜로 쓰는 구글의 Gmail이 10G가 넘는다.
우정훈( Columbia medical school, Biomedical informatics PhD course & Geference Cofounder )의 의견
또하나의 가능성은 Genome sequencing 자체 가격이 떨어져, 마치 병원에서 필요할 때 마다 X-ray 를 찍듯, one time test 로 변모할 가능성이다. Whole genome sequencing 가격이 $1000 아래로 떨어진다면, Genome sequencing 이 필요할 때 마다 새로이 분석을 해도 하등의 문제가 없다 ( 지금 당장 MRI를 생각해 보면 명확하다). 이렇게 되면 굳이 raw data를 어딘가에 저장해 둘 필요가 없으면서, 미래의 분석 능력 향상 때 raw data를 새로이 분석해서 이용해야할 필요성이 완전히 사라지게 된다. 사실 이 시나리오가 가장 현실에 가까울 것 같다.
2. Genome 해석의 불완전성 으로 인한 책임 문제
문제 개괄
현재 Genome의 분석 과정에는 엄청나게 많은 error / variation 이 존재한다. 각 단계 마다 수많은 processing 방법들 중 하나를 선택할 수 있고, 이용할 수 있는 무수히 많은 소프트웨어들이 존재한다. 이들 방법론/소프트웨어들은 상당 부분 상이한 결과를 도출하는 경우가 많다. 잘못된 분석 단계 / 소프트웨어 이용으로 잘못된 결과를 도출하고 이에 기반하여 환자에게 잘못된 치료를 하게되는 문제 상황이 발생할 수 있다.
이종극 박사님, 최형진 선생님 의견
환자를 진료하는 의사 입장에서 여러가지 lab test 들과 진료 장비들을 일상적으로 사용하지만, 이들의 error율도 상당하다. 환자 치료에는 그 시점에서 state-of-art 검사 장비와 test 들을 이용하지만, 현재의 이런 최신 기법들을 훗날 돌아보면 모두 허점들이 있을 수밖에 없다. 하지만, 현재 시점에서 '최선'의 방법들을 선택할 수밖에 없는 것이고, 여기에 설령 error가 들어 있다손 치더라도, 이는 감수할 수 밖에 없는 부분이다.
Genome 의 이용도 이와 같은 맥락에서 바라볼 수 있다고 본다. 현 시점에서 개인의 Genome 분석에 error가 들어 있더라도, 이것이 현재 할 수 있는 최선이었다면, 그것으로 된 것이고, 추후 Genome 분석 방법이 upgrade 되어 error가 발견된다고 하더라도 이는 감수해야 하는 부분이다.
개인적 첨언
두분의 의견에 전적으로 동의하고, 현재 미국에서 이와 관련된 문제들에 어떻게 대처하고 있는지 현재 상황들의 정리해 본다.
Genome 치료의 임상 적용 사례중 가장 잘 알려진 사례는 스티브 잡스 건. 잡스의 개인 유전체 분석에는 지구상 최고의 의료팀이라 해도 과언이 아닌 드림팀이 구성되어 참여 했었고, 당연히 그 당시 잡스 유전체 분석에 State-of-art best practice가 적용되었다. 잡스는 게놈 분석을 위해서만 1억원의 비용을 지불했다. 의료진은 최선의 분석을 하고 치료에 이용했지만, 뚜렷한 치료법을 찾지 못했고 잡스는 사망했다. 잡스의 유전체 분석이 2010년경 행해졌기 때문에, 2년이 지난 지금 시점에서 다시 잡스의 게놈을 분석하면 분명 그 때에는 발견하지 못했던 어쩌면 잡스를 살릴 수도 있었던 중요한 내용을 발견할 가능성도 있다.
하지만, 지금 시점에서 잡스 게놈의 분석을 문제 삼는 사람은 아무도 없다. 잡스의 게놈은 치료가 필요한 바로 그 '시점'에서 필요한 정보였고, 그 당시 이를 최대한 활용했다면, 그것으로 된 것이고, 여기에 법적/도덕적 책임을 지을 하등의 이유가 없다.
잡스나 와트맨 같은 사례들이 나오고, Cancer 환자들에 Genome 분석을 이용하는 Foundation medicine 같은 회사가 구글/빌게이츠 등의 5천억 투자를 받아가며 성공적으로 비즈니스를 런칭했고, Drug 처방에 국한되는 Foundation medicine 보다 한발 더 나아가, 의사의 진료 현장에서 clinical decision 에 적극적으로 개입하는 '예측 모델'을 기반으로한 SV Bio 같은 회사들이 설립되고, 실제 뚜렷한 진단 부터가 어려운 희귀병 환자들을 우선적인 타겟으로 하여 비즈니스를 시작하고 있다.
현재의 시장을 관찰해 보면 개인 Genome의 의료계 도입은 현재 현대 의학으로 뚜렷한 답이 없는 부분에서 부터 적극적으로 도입되고 있는 것을 알 수 있다. 스티브 잡스와 같은 말기 암 화자, 루카스 와트맨 처럼 모든 가능한 치료법을 시도해 봤지만 실패하고 희망이 없는 암 환자, 병명도 모르는 희귀병 환자들.
Genome으로 인한 진단과 처방, 치료는 지금 상황에서 '이것이 정답' 이라고 얘기할 수 있는 임상 적용 사례는 몇몇 Drug에 대한 약물 반응성( Pharmacogenomics ) 분석 등 소수에 불과하다. 하지만, Genome 분석의 도입이 진단과 치료에 의미 있는 한 차원의 데이터를 더해 주고, 이를 기반으로 조금 더 정확하고, 조금 더 정밀한 진단과 치료가 가능해 지고, 또한 개선되어 나갈 것이라고 생각한다.
모임의 말미에 아산병원 신수용 선생님이 개인 게놈 정보를 실제 병원에서 의학적으로 이용함에 있어 해결해야할 굉장히 중요한 문제점들을 제시해 주셨고, 이에 대해 다양한 시각의 의견들이 제시가 되었다. 흥미로운 주제이기도 하고, 또 앞으로 Genome의 산업적 이용에 있어 매우 중요한 사안들이기에, 이렇게 글로 정리하여 많은 사람들과 나누는 것이 의미 있을 거라는 판단에 정리해 본다.
( 기억력의 한계로 제대로 정리되지 않은 부분이 포함되었을 수 있고 상당 부분 논의된 내용이 빠져 있고, 개인적으로 인상 깊었던 두가지 사안에 대해서만 정리를 했습니다. 혹, 잘못된 부분/추가/수정 해야할 부분이 있으면 지적해 주시면 감사하겠습니다 )
1. 개인 게놈 raw data 의 저장 문제
문제 개괄
현재 NGS machine에서 한 사람의 whole human genome을 30x 로 읽으면 fastq raw data size는 180GB(3GB*30*2 -for fastq quality score space- )가 넘어가는 Big Data가 생성되고, 이를 기반으로 많은 환자들의 치료에 이용하는 병원과 같은 곳의 Genome center는 심각한 data storage 문제가 발생한다. 흔히 이에 대한 해결책으로 얘기되고 있는 Cloud 시스템은 a) 이용 요금이 생각보다 싸지 않고, b) 한 사람의 raw data 전송에 수일이 소요되는 전송 속도의 문제 등으로 인하여 적절한 해결책이 되지 못할 공산이 크다.
이종극 박사님 의견
병원에서 raw data를 계속 저장하고 있을 필요가 없다. 데이터 분석에 이용되는 환경( reference genome 과 분석에 이용되는 DB 들의 update )의 변화로 설령 추후에 분석 내용이 변경될 수 있다고 하더라도, 병원에서 genome data의 효용은 환자를 치료하는 시점( at the point of care )에 국한되고, 이 시점이 지난 시점은 이미 데이터 효용이 사라진 후기 때문에 raw data를 가지고 있을 필요가 없다.
최형진 선생님( 충북대 내분비내과 ) 의견
현장의 의사는 현실적으로 개개의 환자를 진료하는데 많은 시간을 쏟을 수 없다. 의사에게 전달되는 Genome 분석 결과는 한두페이지 짜리 최종 분석 보고서면 충분하다. 또한 현재 환자 치료에 이 분석 내용을 활용하였다면, 추후 다시 이 환자의 genome raw data를 재분석해 활용해 얻는 이익은 미미할 것이고, 현실적으로도 이런 재분석이 필요한 경우는 흔하지 않을 것이다.
결론적으로 raw data 저장에 여러가지 현실적 제약이 있다면, 병원이 raw data를 계속 가지고 있을 필요는 없다고 본다.
개인적 의견
Genome raw data storage는 기술의 발전이 자연스럽게 해결해 줄 것이라고 생각한다. 사실 현재의 raw data 사이즈가 큰 이유는 NGS 기계들의 떨어지는 성능 탓이다. DNA 30억개 문자로 3G면 되는 개인 게놈을 왜 30X 즉 30배로 읽을까? a) NGS 기계들이 200 base 단위 정도의 짧은 조각으로 게놈을 읽고, b) 그 마저도 1% 정도의 에러가 섞이기 때문이다. 그래서 전체 게놈의 30배 정도로 200bp 길이의 짧은 조각 DNA read 들을 생산해 '퍼즐 맞추기' 과정을 통해 문제 a 를 해결하고, 같은 부분에 읽힌 30개의 DNA를 확인해 b 문제를 해결하는 형태로 현재의 NGS 기계들이 작동하고 있기 때문이다.
현재 내부적으로 fine-tuning 과정을 거치고 있는 Oxford nanopore 의 4세대 DNA sequencer 가 그들의 주장대로 기계의 성능을 갖춰 출시된다면, 개인 게놈 raw data 사이즈는 혁신적으로 작아지게 된다. Oxford 의 read 길이는 평균 1만 base ( 2012년 초 기준) , error rate 4%( 역시 2012년 초 기준) 다. Oxford는 2012년말 보도 자료에서 error rate 이 1% 이내가 되기 전에는 시장에 출시하지 않겠다고 발표했다. 즉, 시장에 나오는 Oxford 제품은 평균 read 길이 최소 1만에 1% 이하의 error rate의 read를 생산하는 획기적인 DNA sequencer 가 된다.
Oxford 이후에도 Sequencer 성능은 꾸준히 개선될 것이고, 일정 시점에선 whole genome을 그냥 한번 읽어내면 되는 Sequencer 완성형 제품이 나오게 될 것이라고 생각한다. 그렇게 되면 그냥 개인 게놈은 3G 가 된다. 3G 라면 raw data 저장 문제도 그냥 해결이 된다. 현재 우리가 공짜로 쓰는 구글의 Gmail이 10G가 넘는다.
우정훈( Columbia medical school, Biomedical informatics PhD course & Geference Cofounder )의 의견
또하나의 가능성은 Genome sequencing 자체 가격이 떨어져, 마치 병원에서 필요할 때 마다 X-ray 를 찍듯, one time test 로 변모할 가능성이다. Whole genome sequencing 가격이 $1000 아래로 떨어진다면, Genome sequencing 이 필요할 때 마다 새로이 분석을 해도 하등의 문제가 없다 ( 지금 당장 MRI를 생각해 보면 명확하다). 이렇게 되면 굳이 raw data를 어딘가에 저장해 둘 필요가 없으면서, 미래의 분석 능력 향상 때 raw data를 새로이 분석해서 이용해야할 필요성이 완전히 사라지게 된다. 사실 이 시나리오가 가장 현실에 가까울 것 같다.
2. Genome 해석의 불완전성 으로 인한 책임 문제
문제 개괄
현재 Genome의 분석 과정에는 엄청나게 많은 error / variation 이 존재한다. 각 단계 마다 수많은 processing 방법들 중 하나를 선택할 수 있고, 이용할 수 있는 무수히 많은 소프트웨어들이 존재한다. 이들 방법론/소프트웨어들은 상당 부분 상이한 결과를 도출하는 경우가 많다. 잘못된 분석 단계 / 소프트웨어 이용으로 잘못된 결과를 도출하고 이에 기반하여 환자에게 잘못된 치료를 하게되는 문제 상황이 발생할 수 있다.
이종극 박사님, 최형진 선생님 의견
환자를 진료하는 의사 입장에서 여러가지 lab test 들과 진료 장비들을 일상적으로 사용하지만, 이들의 error율도 상당하다. 환자 치료에는 그 시점에서 state-of-art 검사 장비와 test 들을 이용하지만, 현재의 이런 최신 기법들을 훗날 돌아보면 모두 허점들이 있을 수밖에 없다. 하지만, 현재 시점에서 '최선'의 방법들을 선택할 수밖에 없는 것이고, 여기에 설령 error가 들어 있다손 치더라도, 이는 감수할 수 밖에 없는 부분이다.
Genome 의 이용도 이와 같은 맥락에서 바라볼 수 있다고 본다. 현 시점에서 개인의 Genome 분석에 error가 들어 있더라도, 이것이 현재 할 수 있는 최선이었다면, 그것으로 된 것이고, 추후 Genome 분석 방법이 upgrade 되어 error가 발견된다고 하더라도 이는 감수해야 하는 부분이다.
개인적 첨언
두분의 의견에 전적으로 동의하고, 현재 미국에서 이와 관련된 문제들에 어떻게 대처하고 있는지 현재 상황들의 정리해 본다.
Genome 치료의 임상 적용 사례중 가장 잘 알려진 사례는 스티브 잡스 건. 잡스의 개인 유전체 분석에는 지구상 최고의 의료팀이라 해도 과언이 아닌 드림팀이 구성되어 참여 했었고, 당연히 그 당시 잡스 유전체 분석에 State-of-art best practice가 적용되었다. 잡스는 게놈 분석을 위해서만 1억원의 비용을 지불했다. 의료진은 최선의 분석을 하고 치료에 이용했지만, 뚜렷한 치료법을 찾지 못했고 잡스는 사망했다. 잡스의 유전체 분석이 2010년경 행해졌기 때문에, 2년이 지난 지금 시점에서 다시 잡스의 게놈을 분석하면 분명 그 때에는 발견하지 못했던 어쩌면 잡스를 살릴 수도 있었던 중요한 내용을 발견할 가능성도 있다.
하지만, 지금 시점에서 잡스 게놈의 분석을 문제 삼는 사람은 아무도 없다. 잡스의 게놈은 치료가 필요한 바로 그 '시점'에서 필요한 정보였고, 그 당시 이를 최대한 활용했다면, 그것으로 된 것이고, 여기에 법적/도덕적 책임을 지을 하등의 이유가 없다.
잡스나 와트맨 같은 사례들이 나오고, Cancer 환자들에 Genome 분석을 이용하는 Foundation medicine 같은 회사가 구글/빌게이츠 등의 5천억 투자를 받아가며 성공적으로 비즈니스를 런칭했고, Drug 처방에 국한되는 Foundation medicine 보다 한발 더 나아가, 의사의 진료 현장에서 clinical decision 에 적극적으로 개입하는 '예측 모델'을 기반으로한 SV Bio 같은 회사들이 설립되고, 실제 뚜렷한 진단 부터가 어려운 희귀병 환자들을 우선적인 타겟으로 하여 비즈니스를 시작하고 있다.
현재의 시장을 관찰해 보면 개인 Genome의 의료계 도입은 현재 현대 의학으로 뚜렷한 답이 없는 부분에서 부터 적극적으로 도입되고 있는 것을 알 수 있다. 스티브 잡스와 같은 말기 암 화자, 루카스 와트맨 처럼 모든 가능한 치료법을 시도해 봤지만 실패하고 희망이 없는 암 환자, 병명도 모르는 희귀병 환자들.
Genome으로 인한 진단과 처방, 치료는 지금 상황에서 '이것이 정답' 이라고 얘기할 수 있는 임상 적용 사례는 몇몇 Drug에 대한 약물 반응성( Pharmacogenomics ) 분석 등 소수에 불과하다. 하지만, Genome 분석의 도입이 진단과 치료에 의미 있는 한 차원의 데이터를 더해 주고, 이를 기반으로 조금 더 정확하고, 조금 더 정밀한 진단과 치료가 가능해 지고, 또한 개선되어 나갈 것이라고 생각한다.
Saturday, November 17, 2012
인간게놈프로젝트(Human Genome project)와 생물정보학(Bioinformatics)
Bioinformatics가 언론의 통해 기사화 되고 널리 알려지기 시작한 계기는 Genome Project 의 막바지인 1990년대 후반이 되면서 부터 였다. 인간게놈프로젝트( HGP: Human Genome Project)라 알려진 이 프로젝트는 10년의 시간에 걸쳐 미국 주도의 다국적 연구 컨소시움에 의해 총 30억달러 우리돈 3조원 이상이 소모된 달탐사 프로젝트 이래 최대의 과학 프로젝트 였다.
하지만, 1990년대 후반 크레이그 벤터( J. Craig Venter )가 셀레라(Celera genomics)사를 설립 대규모 자동 게놈 해독기를 장착하고 shortgun sequencing이라는 초고속 게놈 해독법을 이용해 인간 게놈을 분석하면서 이 지루한 프로젝트는 흥미진진한 소설 같은 상황으로 전개가 된다.
셀레라의 분석 기술로는 정부 주도의 다국적 팀에 의해 10년 이상이 소요될 것으로 예측되는 프로젝트를 불과 2-3년 안에 완료 할 수 있었고, 미생물 게놈을 빠른 시간 안에 분석 해 발표하면서 이는 기정 사실화 되었다. 정부 주도 하의 다국적 팀에게 이는 엄청난 위협이었고, 위기감을 불러 일으켰다. ( 과학자의 입장에서 보자면 수년간 노력한 일이 수포로 돌아가는 것과 마찬가지인데, 과학 연구에서 2등은 큰 의미가 없기 때문. 이와 관련한 스토리는 벤터의 자서전 Life decoded에 자세히 기록되어 있는데, 정부 주도 팀의 관료적 행태들은 구역질이 날 정도의 장면들이 많다. )
Shortgun genome sequencing
Shortgun sequencing 은 인간의 긴 30억개 염기 서열을 평균 500-1,000 개 염기 길이의 작은 단위로 잘라 읽은 후, 각각을 퍼즐 맞추기 식으로 이어 붙여 전체 30억개 염기 서열 부위를 완성하는 형태다.
Shotgun sequencing 방법은 얼핏 매우 간단해 보이지만, 이 방법은 Bioinformatics 가 없이는 애초에 불가능한 서열 분석 방법이었다.
Shortgun sequencing을 통해 예를 들어 세개의 염기 서열 조각이 아래와 같이 주어진다고 하면 어떻게 이어 붙여야 할까?
AGTGT, GTAAC, ACTAG
아래와 같이 이어 붙이면 된다.
AGTGT
GTAAC
ACTAG
--------------------------
AGTGTAACTAG
아래와 같은 방식도 가능하다.
ACTAG
AGTGT
GTAAC
--------------------------
ACTAGTGTAAC
어떤 쪽이 옳은 것일까? 이런 서열 조각이 수천만개 있다면 어떨까? 어떻게 이 조각들을 제대로 꿰어 맞출 수 있을까? 이는 굉장히 정교한 수학적 모델을 필요로 했으며, 엄청난 컴퓨팅 파워를 필요로 하는 문제로 정통 생물학자들이 해결할 수 있는 문제가 아니었다.
이 문제를 푸는데 적용할 만한 수학적 이론은 Shortest common superstring problem (SSP), Eulerian graph problem 등이 있었고, Genome sequencing에 적용할 만한 이론들로 Michael Waterman 등의 기여로 대체로 정립,개발되어 왔지만, 실제로 이를 대량의 염기 서열 조각을 이어 붙이는 시스템에 적용하여 제작된 전례가 없었다.
생물정보학자 전쟁 :: Eugene Myers vs. Jim Kent
Eugene Myers 를 필두로 하는 Bioinformatics 팀이 셀레라 측의 Shortgun sequencing을 위한 염기 서열 조각 맞추기 문제를 위한 시스템을 성공적으로 완성했다. Gene Myers라고 흔히 불리는 Eugene Myers 는 NCBI에서 1990년 지금까지 가장 광범위하고 성공적으로 사용되는 생물학 프로그램 BLAST 를 공동 개발한 Bioinformatics 초기 영웅 중 한명이었는데, 그가 Celera에 합류하여 라떼 커피를 친구삼아 밤낮 가리지 않고 일에 몰두한 덕에 비소로 셀레라의 Shortgun sequencing 기술이 완성될 수 있었다.
Shortgun sequencing 시스템을 하드웨어적, 소프트웨어적으로 완벽히 갖추고 인간 게놈 분석의 막바지에 다다르고 있었던 셀레라 사에 비해, 다국적팀( 인간게놈컨소시움)은 정부의 중재로 셀레라 사와 함께 인간 게놈 분석 초안을 발표하기로 약속한 몇달 전이 되기 까지 이 게놈 조각 퍼즐 맞추기( Sequence assembly ) 를 위한 시스템이 완성되지 않아 초조하게 발을 구르는 상황이었다.
10년의 노력, 3조원의 정부 연구비를 정당화 하기 위해서 이 시스템은 너무나 간절했던 것이었다. 이 때, UCSC 생물학과에서 박사과정 학생이던 Jim Kent (William James Kent )가 David Haussler 와의 협력 하에 학교에서 새로 구매한 50대의 컴퓨터를 리눅스 클러스터로 사용하여 한달 만에 이 시스템을 완성한다. Haussler와 협력이라고 하지만, 이 프로그램의 코드는 100% Kent 혼자서 한달 동안 밤낮없이 달려들어 완성한 것이었다. Kent가 완성한 이 프로그램을 이용해 비로소 다국적팀도 셀레라사에 뒤지지 않게 성공적으로 전체 인간 게놈 지도를 완성할 수 있었다. ( 정확히는 Kent의 프로그램을 이용한 다국적 팀이 3일 빨리 완성)
Kent가 생물학과 박사과정 학생이었지만, 사실 그는 3D graphics 엔진을 개발하는 컴퓨터 프로그래머로 일하던 전문 프로그래머였는데, 생물학에 매력을 느껴 community college에서 생물학 과목을 수강한 후, UCSC 생물학과 박사과정에 늦깍이 진학을 한 특이한 이력을 가지고 있던 학생이었다.
이 때의 성과를 바탕으로 UCSC는 UCSC genome browser라는 웹 기반 게놈 검색/분석 시스템을 서비스하게 되는데, 현재 이는 BLAST와 마찬가지로 생물학자들에게 매우 일상적으로 이용되는 Bioinformatics 서비스로 자리 메김했고, 덩달아 UCSC 는 그저그런 UC 계열 대학에서 세계 최정상의 게놈 연구 기관 중 한 곳으로 자리 메김 하게 되었다.
( Kent의 사례는 왜 다양한 background를 가진 직원/학생들을 창의적 업무를 요하는 회사/대학에서 뽑아야 하는지를 극명하게 보여주는 또하나의 좋은 예가 된다 )
Bioinformatics on the go
Bioinformatics 라는 분야는 Computational biology 라고 명명된 좀 더 수학적 이론에 치우친 분야를 토대로 게놈프로젝트 이전 1970년대 부터 이미 존재해 온 분야였지만, 인간게놈프로젝트를 기점으로 그 필요성과 역할이 크게 확대되었으며, 이에 대한 수요도 크게 늘어나게 되었다.
인간게놈프로젝트를 기점으로 비로소 독립된 Bioinformatics 교육 프로그램들이 미국의 대학들에 설립되기 시작했고( 연구 조직이 있었던 곳은 꾀나 있었지만, 독립적 학위 과정 프로그램을 가진 곳은 없었다), Michael Waterman이 1982년 부터 그 기틀을 잡아왔던 USC, 인간게놈프로젝트를 통해 유명해진 David Haussler 가 중심이 된 UCSC, 그외 Boston University 가 초기 독립된 Bioinformatics 학위 과정을 대학에 개설했는데, 이중 UCSC는 학부생 레벨( Bachelor degree)의 코스를 유일하게 설립해 운영을 시작했다.
국내에서는 2001년 숭실대학교에 최초의 학부 과정 학과가 개설(현재는 의생명공학부)되었고, 부산대학에 최초의 대학원 과정이 개설되었다. 2002년에는 서울대에 생물정보협동과정 대학원 과정이 개설되었고, 2003년에는 정문술 회장의 300억 기부로 KAIST에 Biosystems 이라는 이름으로 Bioinformatics 를 교육하는 학과(현재는 Bio & Brain Engineering 학과)가 만들어졌다.
인간게놈프로젝트에서 극명하게 드러난 것은 생물학이 이제 Large scale data 생산하는 시대에 접어들었고, 생물학이 이 large scale data의 분석을 통해 연구되는 Information science 즉 정보과학의 시대로 접어들었다는 사실이었다.
이는 게놈프로젝트 이후 생물학의 트렌드를 보면 극명히 드러나는데, 이 후에 생물학의 세부 분야들은 Genome 처럼 총체라는 뜻을 더해주는 -ome이 붙는 High-Thoughput data science 로 이름이 붙여졌고 ( Exressome, Proteome, Interactome, Metabolom , etc ) , 뛰어난 생물학 연구 팀에는 의례히 뛰어난 Bioinformatics 인력 혹은 공동연구팀이 함께 연구를 이끌어 가고 있음을 확인할 수 있다. 다시 말하면, 이제 첨단 생물학을 연구하기 위해서는 bioinformatics 로 대변되는 정보 과학적 분석 능력이 필수가 되었다는 말이 된다.
최근에 게놈 분석 비용이 획기적으로 낮아지면서, 게놈의 대중화와 이와 연계된 산업적 폭발이 목전에 와 있는데, 여기서도 게놈의 분석/관리/서비스를 위한 Bioinformatics 인력이 주도적 역할을 하게될 터인데,
게놈프로젝트 이후 많은 대학에 학위 과정이 개설되어 많은 인력을 배출해낸 미국에 비해 한국은 그나마 교육과정을 개설했던 학교들에서 오히려 Bioinformatics 이외의 분야들로 타겟을 바꾸어 학과를 포지셔닝 하면서, 제대로된 Bioinformatics 교육 커리큘럼을 갖춘 학교가 전무한 실정이다. 사실 미국을 제외하면 Bioinformatics 학위 과정이 활성화된 나라가 거의 없긴 한데, 그래서 자연히 앞으로의 Bioinformatics 분야도 미국이 주도적으로 발전시켜 나갈 수밖에 없지 않나 생각한다.
여담으로 현재 국내에서 중급 Bioinformatics 인력 조차도 구하기 쉽지 않은데, 앞으로 꾀나 오랜 시간 이런 흐름이 지속되지 않을가 조심스레 예측해 본다.
하지만, 1990년대 후반 크레이그 벤터( J. Craig Venter )가 셀레라(Celera genomics)사를 설립 대규모 자동 게놈 해독기를 장착하고 shortgun sequencing이라는 초고속 게놈 해독법을 이용해 인간 게놈을 분석하면서 이 지루한 프로젝트는 흥미진진한 소설 같은 상황으로 전개가 된다.
셀레라의 분석 기술로는 정부 주도의 다국적 팀에 의해 10년 이상이 소요될 것으로 예측되는 프로젝트를 불과 2-3년 안에 완료 할 수 있었고, 미생물 게놈을 빠른 시간 안에 분석 해 발표하면서 이는 기정 사실화 되었다. 정부 주도 하의 다국적 팀에게 이는 엄청난 위협이었고, 위기감을 불러 일으켰다. ( 과학자의 입장에서 보자면 수년간 노력한 일이 수포로 돌아가는 것과 마찬가지인데, 과학 연구에서 2등은 큰 의미가 없기 때문. 이와 관련한 스토리는 벤터의 자서전 Life decoded에 자세히 기록되어 있는데, 정부 주도 팀의 관료적 행태들은 구역질이 날 정도의 장면들이 많다. )
Shortgun genome sequencing
Shortgun sequencing 은 인간의 긴 30억개 염기 서열을 평균 500-1,000 개 염기 길이의 작은 단위로 잘라 읽은 후, 각각을 퍼즐 맞추기 식으로 이어 붙여 전체 30억개 염기 서열 부위를 완성하는 형태다.
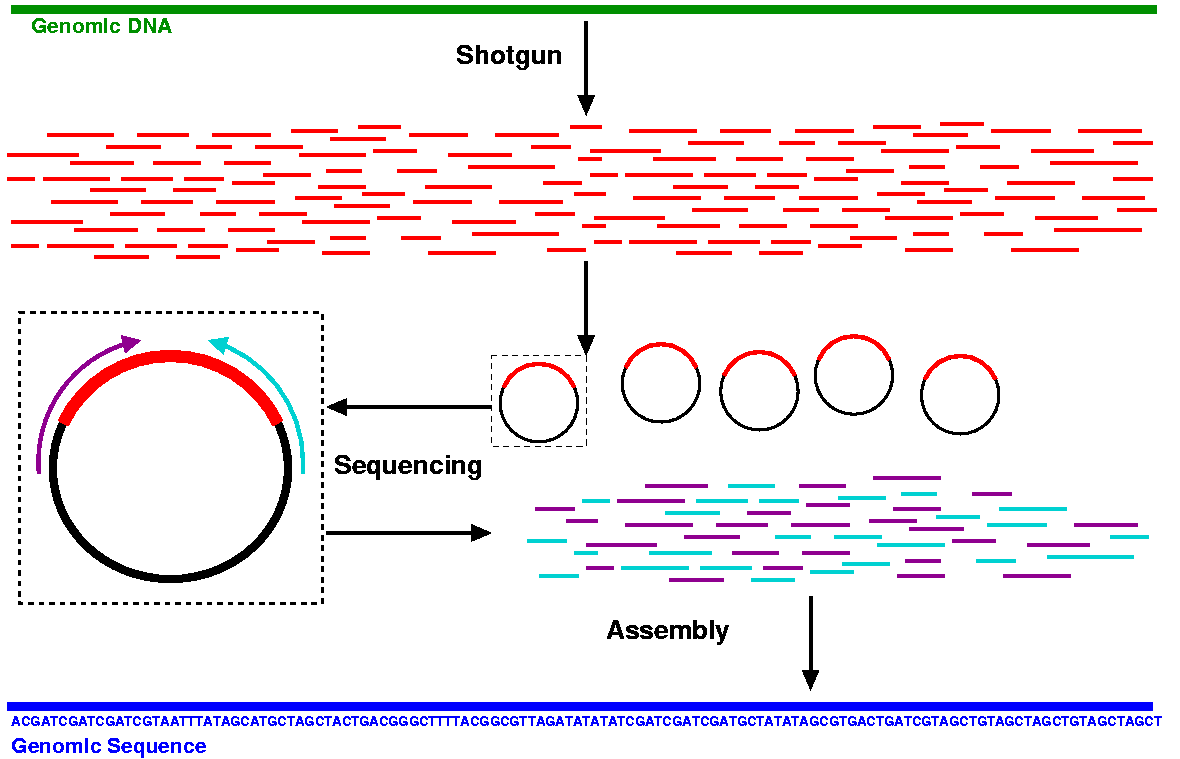 |
| Shortgun sequencing 방법 모식도 |
Shotgun sequencing 방법은 얼핏 매우 간단해 보이지만, 이 방법은 Bioinformatics 가 없이는 애초에 불가능한 서열 분석 방법이었다.
Shortgun sequencing을 통해 예를 들어 세개의 염기 서열 조각이 아래와 같이 주어진다고 하면 어떻게 이어 붙여야 할까?
AGTGT, GTAAC, ACTAG
아래와 같이 이어 붙이면 된다.
AGTGT
GTAAC
ACTAG
--------------------------
AGTGTAACTAG
아래와 같은 방식도 가능하다.
ACTAG
AGTGT
GTAAC
--------------------------
ACTAGTGTAAC
어떤 쪽이 옳은 것일까? 이런 서열 조각이 수천만개 있다면 어떨까? 어떻게 이 조각들을 제대로 꿰어 맞출 수 있을까? 이는 굉장히 정교한 수학적 모델을 필요로 했으며, 엄청난 컴퓨팅 파워를 필요로 하는 문제로 정통 생물학자들이 해결할 수 있는 문제가 아니었다.
이 문제를 푸는데 적용할 만한 수학적 이론은 Shortest common superstring problem (SSP), Eulerian graph problem 등이 있었고, Genome sequencing에 적용할 만한 이론들로 Michael Waterman 등의 기여로 대체로 정립,개발되어 왔지만, 실제로 이를 대량의 염기 서열 조각을 이어 붙이는 시스템에 적용하여 제작된 전례가 없었다.
생물정보학자 전쟁 :: Eugene Myers vs. Jim Kent
Eugene Myers 를 필두로 하는 Bioinformatics 팀이 셀레라 측의 Shortgun sequencing을 위한 염기 서열 조각 맞추기 문제를 위한 시스템을 성공적으로 완성했다. Gene Myers라고 흔히 불리는 Eugene Myers 는 NCBI에서 1990년 지금까지 가장 광범위하고 성공적으로 사용되는 생물학 프로그램 BLAST 를 공동 개발한 Bioinformatics 초기 영웅 중 한명이었는데, 그가 Celera에 합류하여 라떼 커피를 친구삼아 밤낮 가리지 않고 일에 몰두한 덕에 비소로 셀레라의 Shortgun sequencing 기술이 완성될 수 있었다.
Shortgun sequencing 시스템을 하드웨어적, 소프트웨어적으로 완벽히 갖추고 인간 게놈 분석의 막바지에 다다르고 있었던 셀레라 사에 비해, 다국적팀( 인간게놈컨소시움)은 정부의 중재로 셀레라 사와 함께 인간 게놈 분석 초안을 발표하기로 약속한 몇달 전이 되기 까지 이 게놈 조각 퍼즐 맞추기( Sequence assembly ) 를 위한 시스템이 완성되지 않아 초조하게 발을 구르는 상황이었다.
10년의 노력, 3조원의 정부 연구비를 정당화 하기 위해서 이 시스템은 너무나 간절했던 것이었다. 이 때, UCSC 생물학과에서 박사과정 학생이던 Jim Kent (William James Kent )가 David Haussler 와의 협력 하에 학교에서 새로 구매한 50대의 컴퓨터를 리눅스 클러스터로 사용하여 한달 만에 이 시스템을 완성한다. Haussler와 협력이라고 하지만, 이 프로그램의 코드는 100% Kent 혼자서 한달 동안 밤낮없이 달려들어 완성한 것이었다. Kent가 완성한 이 프로그램을 이용해 비로소 다국적팀도 셀레라사에 뒤지지 않게 성공적으로 전체 인간 게놈 지도를 완성할 수 있었다. ( 정확히는 Kent의 프로그램을 이용한 다국적 팀이 3일 빨리 완성)
Kent가 생물학과 박사과정 학생이었지만, 사실 그는 3D graphics 엔진을 개발하는 컴퓨터 프로그래머로 일하던 전문 프로그래머였는데, 생물학에 매력을 느껴 community college에서 생물학 과목을 수강한 후, UCSC 생물학과 박사과정에 늦깍이 진학을 한 특이한 이력을 가지고 있던 학생이었다.
이 때의 성과를 바탕으로 UCSC는 UCSC genome browser라는 웹 기반 게놈 검색/분석 시스템을 서비스하게 되는데, 현재 이는 BLAST와 마찬가지로 생물학자들에게 매우 일상적으로 이용되는 Bioinformatics 서비스로 자리 메김했고, 덩달아 UCSC 는 그저그런 UC 계열 대학에서 세계 최정상의 게놈 연구 기관 중 한 곳으로 자리 메김 하게 되었다.
( Kent의 사례는 왜 다양한 background를 가진 직원/학생들을 창의적 업무를 요하는 회사/대학에서 뽑아야 하는지를 극명하게 보여주는 또하나의 좋은 예가 된다 )
Bioinformatics on the go
Bioinformatics 라는 분야는 Computational biology 라고 명명된 좀 더 수학적 이론에 치우친 분야를 토대로 게놈프로젝트 이전 1970년대 부터 이미 존재해 온 분야였지만, 인간게놈프로젝트를 기점으로 그 필요성과 역할이 크게 확대되었으며, 이에 대한 수요도 크게 늘어나게 되었다.
인간게놈프로젝트를 기점으로 비로소 독립된 Bioinformatics 교육 프로그램들이 미국의 대학들에 설립되기 시작했고( 연구 조직이 있었던 곳은 꾀나 있었지만, 독립적 학위 과정 프로그램을 가진 곳은 없었다), Michael Waterman이 1982년 부터 그 기틀을 잡아왔던 USC, 인간게놈프로젝트를 통해 유명해진 David Haussler 가 중심이 된 UCSC, 그외 Boston University 가 초기 독립된 Bioinformatics 학위 과정을 대학에 개설했는데, 이중 UCSC는 학부생 레벨( Bachelor degree)의 코스를 유일하게 설립해 운영을 시작했다.
국내에서는 2001년 숭실대학교에 최초의 학부 과정 학과가 개설(현재는 의생명공학부)되었고, 부산대학에 최초의 대학원 과정이 개설되었다. 2002년에는 서울대에 생물정보협동과정 대학원 과정이 개설되었고, 2003년에는 정문술 회장의 300억 기부로 KAIST에 Biosystems 이라는 이름으로 Bioinformatics 를 교육하는 학과(현재는 Bio & Brain Engineering 학과)가 만들어졌다.
인간게놈프로젝트에서 극명하게 드러난 것은 생물학이 이제 Large scale data 생산하는 시대에 접어들었고, 생물학이 이 large scale data의 분석을 통해 연구되는 Information science 즉 정보과학의 시대로 접어들었다는 사실이었다.
이는 게놈프로젝트 이후 생물학의 트렌드를 보면 극명히 드러나는데, 이 후에 생물학의 세부 분야들은 Genome 처럼 총체라는 뜻을 더해주는 -ome이 붙는 High-Thoughput data science 로 이름이 붙여졌고 ( Exressome, Proteome, Interactome, Metabolom , etc ) , 뛰어난 생물학 연구 팀에는 의례히 뛰어난 Bioinformatics 인력 혹은 공동연구팀이 함께 연구를 이끌어 가고 있음을 확인할 수 있다. 다시 말하면, 이제 첨단 생물학을 연구하기 위해서는 bioinformatics 로 대변되는 정보 과학적 분석 능력이 필수가 되었다는 말이 된다.
최근에 게놈 분석 비용이 획기적으로 낮아지면서, 게놈의 대중화와 이와 연계된 산업적 폭발이 목전에 와 있는데, 여기서도 게놈의 분석/관리/서비스를 위한 Bioinformatics 인력이 주도적 역할을 하게될 터인데,
게놈프로젝트 이후 많은 대학에 학위 과정이 개설되어 많은 인력을 배출해낸 미국에 비해 한국은 그나마 교육과정을 개설했던 학교들에서 오히려 Bioinformatics 이외의 분야들로 타겟을 바꾸어 학과를 포지셔닝 하면서, 제대로된 Bioinformatics 교육 커리큘럼을 갖춘 학교가 전무한 실정이다. 사실 미국을 제외하면 Bioinformatics 학위 과정이 활성화된 나라가 거의 없긴 한데, 그래서 자연히 앞으로의 Bioinformatics 분야도 미국이 주도적으로 발전시켜 나갈 수밖에 없지 않나 생각한다.
여담으로 현재 국내에서 중급 Bioinformatics 인력 조차도 구하기 쉽지 않은데, 앞으로 꾀나 오랜 시간 이런 흐름이 지속되지 않을가 조심스레 예측해 본다.
Subscribe to:
Posts (Atom)
